Kod
source("code/common.R")source("code/common.R")Podstawowym i najbardziej uniwersalnym formatem zapisu danych są pliki tekstowe. Takie pliki mogą przechowywać różnorodne struktury oraz być zapisane w różnorodnych formatach, takich jak CSV, TSV, XML, JSON, YAML, albo zapisane w formacie tekstowym bez żadnego określonego formatu. Najczęściej spotykanym formatem plików tekstowych jest CSV (ang. Comma Separated Values), w którym dane są zapisane w postaci tabeli, gdzie kolumny są oddzielone separatorem, a wiersze są oddzielone znakiem nowej linii. Ten format ma szereg zalet, w tym prostotę, niezmienność, czy możliwość otwarcia w dowolnym edytorze tekstu. Za jako wady można uznać brak standardu tego czym jest separator, brak standardu kodowania znaków, brak możliwości przechowywania metadanych, czy potencjalnie duże rozmiary plików.
library(readr)
gm = read_csv("data/gapminder_1950_2023.csv")
head(gm)# A tibble: 6 × 8
Kraj Rok Region Populacja PKB_na_osobe Ocz_dl_zycia CO2_na_osobe Plodnosc
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Austra… 1950 Azja 8180000 16400 69.4 6.7 3.07
2 Austra… 1951 Azja 8420000 16500 69.2 7.04 3.06
3 Austra… 1952 Azja 8630000 16200 69.5 7.01 3.18
# ℹ 3 more rowsgm2 = read_csv("data/gapminder_1950_2023_ver2.csv")
head(gm2)# A tibble: 6 × 1
`Kraj;Rok;Region;Populacja;PKB_na_osobe;Ocz_dl_zycia;CO2_na_osobe;Plodnosc`
<chr>
1 Australia;1950;Azja;8,18e+06;16400;69,4;6,70000;3,07
2 Australia;1951;Azja;8,42e+06;16500;69,2;7,04000;3,06
3 Australia;1952;Azja;8,63e+06;16200;69,5;7,01000;3,18
# ℹ 3 more rowsgm3 = read_delim("data/gapminder_1950_2023_ver2.csv",
delim = ";",
locale = locale(encoding = "UTF-8", decimal_mark = ","))
head(gm3)# A tibble: 6 × 8
Kraj Rok Region Populacja PKB_na_osobe Ocz_dl_zycia CO2_na_osobe Plodnosc
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Austra… 1950 Azja 8180000 16400 69.4 6.7 3.07
2 Austra… 1951 Azja 8420000 16500 69.2 7.04 3.06
3 Austra… 1952 Azja 8630000 16200 69.5 7.01 3.18
# ℹ 3 more rowsArkusze kalkulacyjne to kolejny sposób przechowywania danych ustrukturyzowanych w postaci plików. Różnią się one od plików tekstowych szeregiem kwestii, takich jak możliwość zapisania wielu arkuszy w jednym pliku czy możliwość zapisania formuł. Ich duża popularność wynika z interaktywności tego podejścia, gdzie użytkownicy mogą wprowadzać dane, modyfikować je, czy też na ich podstawie tworzyć wykresy.
library(readxl)
gm4 = read_excel("data/gapminder_1950_2023.xlsx")
head(gm)# A tibble: 6 × 8
Kraj Rok Region Populacja PKB_na_osobe Ocz_dl_zycia CO2_na_osobe Plodnosc
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Austra… 1950 Azja 8180000 16400 69.4 6.7 3.07
2 Austra… 1951 Azja 8420000 16500 69.2 7.04 3.06
3 Austra… 1952 Azja 8630000 16200 69.5 7.01 3.18
# ℹ 3 more rowsexcel_sheets("data/gapminder_1950_2023.xlsx")[1] "Sheet1"gm5 = read_excel("data/gapminder_1950_2023.xlsx",
sheet = "Sheet1", range = "A1:D10")Sugestie w kwestii organizacji danych w arkuszach kalkulacyjnych: https://doi.org/10.1080/00031305.2017.1375989
Współcześnie ogromne ilości danych przechowywane są w bazach danych. Bazy danych to zbiory danych zorganizowane w systematyczny sposób umożliwiający ich łatwe przechowywanie, zarządzanie, oraz dostęp. Istnieje wiele klasyfikacji systemów zarządzania bazami danych (ang. Database Management System, DBMS). Jedną z możliwości jest rozróżnienie ze względu na miejsce przechowywania, np. lokalne bazy danych (ang., in-process databases) oraz bazy danych klient-serwer (ang. client-server databases). Inne rozróznienie, to podział ze względu na zastosowanie, np. operacyjne bazy danych (ang. Online Transaction Processing, OLTP) oraz analityczne bazy danych (ang. Online Analytical Processing, OLAP). Trzecie przykładowe rozróżnienie to model danych, np. relacyjne bazy danych, bazy danych NoSQL, czy grafowe bazy danych.
Przykładowe systemy zarządzania bazami danych:
library(DBI)
library(dbplyr)library(DBI)
library(RSQLite)
library(dplyr)
conn = dbConnect(RSQLite::SQLite(), "data/gapminder_1950_2023.sqlite")
tables = dbListTables(conn)
print(tables)[1] "gapminder_1950_2023"gm = dbReadTable(conn, "gapminder_1950_2023")
result_sql = dbGetQuery(conn, "SELECT * FROM gapminder_1950_2023 WHERE Rok = 2000")
head(result_sql) Kraj Rok Region Populacja PKB_na_osobe Ocz_dl_zycia CO2_na_osobe
1 Australia 2000 Azja 1.90e+07 40200 79.7 18.500
2 Brunei 2000 Azja 3.34e+05 74300 72.5 13.800
3 Kambodża 2000 Azja 1.21e+07 1590 60.4 0.163
4 Chiny 2000 Azja 1.26e+09 3800 71.3 2.600
5 Fidżi 2000 Azja 8.33e+05 9360 65.8 1.030
6 Hongkong, Chiny 2000 Azja 6.73e+06 37700 80.4 6.100
Plodnosc
1 1.76
2 2.22
3 3.81
4 1.50
5 3.09
6 0.98# Wykonanie zapytania dbplyr
gm_db = tbl(conn, "gapminder_1950_2023")
gm_db_filter = filter(gm_db, Rok == 2000)
gm_db_filter# Source: SQL [?? x 8]
# Database: sqlite 3.45.2 [/home/jakub/teaching/auto-wiz-danych/data/gapminder_1950_2023.sqlite]
Kraj Rok Region Populacja PKB_na_osobe Ocz_dl_zycia CO2_na_osobe Plodnosc
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Austra… 2000 Azja 19000000 40200 79.7 18.5 1.76
2 Brunei 2000 Azja 334000 74300 72.5 13.8 2.22
3 Kambod… 2000 Azja 12100000 1590 60.4 0.163 3.81
# ℹ more rowsgm_db_collect = collect(gm_db_filter)
gm_db_collect# A tibble: 190 × 8
Kraj Rok Region Populacja PKB_na_osobe Ocz_dl_zycia CO2_na_osobe Plodnosc
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Austra… 2000 Azja 19000000 40200 79.7 18.5 1.76
2 Brunei 2000 Azja 334000 74300 72.5 13.8 2.22
3 Kambod… 2000 Azja 12100000 1590 60.4 0.163 3.81
# ℹ 187 more rowsdbDisconnect(conn)conn = dbConnect(RSQLite::SQLite(), "data/gapminder_1950_2023.sqlite")
gm_db = tbl(conn, "gapminder_1950_2023")
filter(gm_db, Rok == 2000) |>
show_query()<SQL>
SELECT `gapminder_1950_2023`.*
FROM `gapminder_1950_2023`
WHERE (`Rok` = 2000.0)dbDisconnect(conn)Jak wspomniano wcześniej, pliki tekstowe mogą przechowywać różnorodne struktury danych. Jednakże, w przypadku bardziej złożonych struktur, takich jak dane geoprzestrzenne, serie czasowe, czy dane hierarchiczne, proste pliki tekstowe (np. CSV) mogą okazać się niewystarczające. W takich przypadkach często stosuje się formaty częściowo uporządkowane, takie jak XML czy JSON.
Format XML (ang. eXtensible Markup Language) jest językiem znaczników. Jednym z jego głównych założeń jest to, że możliwe jest tworzenie własnych znaczników, co pozwala na tworzenie bardziej złożonych i dopasowanych do konkretnych potrzeb struktur danych.
Format XML składa się z elementów, które mogą zawierać atrybuty, tekst, inne elementy (zagnieżdzenia), czy też być puste. Elementy w XMLu oznacza się za pomocą znaczników, które składają się z nazwy elementu oraz opcjonalnych atrybutów, np. <element atrybut="wartość"/>.
QGIS przechowuje style warstw w formacie XML o rozszerzeniu .qml. Poniżej widać początek pliku clc_legend_qgis_raster.qml, który przechowuje legendę dla warstwy rastrowej opisującej zbiór danych Corine Land Cover.
<!DOCTYPE qgis PUBLIC 'http://mrcc.com/qgis.dtd' 'SYSTEM'>
<qgis minScale="1e+08" version="3.4.12-Madeira" styleCategories="AllStyleCategories" maxScale="0" hasScaleBasedVisibilityFlag="0">
<flags>
<Identifiable>1</Identifiable>
<Removable>1</Removable>
<Searchable>1</Searchable>
</flags>
<customproperties>
<property value="false" key="WMSBackgroundLayer"/>
...
</customproperties>
...
</qgis>Taki format możemy wczytać, przetwarzać, a także zapisywać za pomocą pakietu R xml2. W poniższym przykładzie interesuje nas wydobycie kolorów oraz etykiet z pliku clc_legend_qgis_raster.qml.
library(xml2)
qgis_legend = read_xml("data/clc_legend_qgis_raster.qml")
qgis_legend{xml_document}
<qgis minScale="1e+08" version="3.4.12-Madeira" styleCategories="AllStyleCategories" maxScale="0" hasScaleBasedVisibilityFlag="0">
[1] <flags>\n <Identifiable>1</Identifiable>\n <Removable>1</Removable>\n ...
[2] <customproperties>\n <property value="false" key="WMSBackgroundLayer"/>\ ...
[3] <pipe>\n <rasterrenderer opacity="1" alphaBand="-1" band="1" type="palet ...
[4] <blendMode>0</blendMode>nodes = xml_find_all(qgis_legend, "//paletteEntry")
nodes{xml_nodeset (45)}
[1] <paletteEntry color="#e6004d" label="111 - Continuous urban fabric" valu ...
[2] <paletteEntry color="#ff0000" label="112 - Discontinuous urban fabric" v ...
[3] <paletteEntry color="#cc4df2" label="121 - Industrial or commercial unit ...
[4] <paletteEntry color="#cc0000" label="122 - Road and rail networks and as ...
[5] <paletteEntry color="#e6cccc" label="123 - Port areas" value="5" alpha=" ...
[6] <paletteEntry color="#e6cce6" label="124 - Airports" value="6" alpha="25 ...
[7] <paletteEntry color="#a600cc" label="131 - Mineral extraction sites" val ...
[8] <paletteEntry color="#a64d00" label="132 - Dump sites" value="8" alpha=" ...
[9] <paletteEntry color="#ff4dff" label="133 - Construction sites" value="9" ...
[10] <paletteEntry color="#ffa6ff" label="141 - Green urban areas" value="10" ...
[11] <paletteEntry color="#ffe6ff" label="142 - Sport and leisure facilities" ...
[12] <paletteEntry color="#ffffa8" label="211 - Non-irrigated arable land" va ...
[13] <paletteEntry color="#ffff00" label="212 - Permanently irrigated land" v ...
[14] <paletteEntry color="#e6e600" label="213 - Rice fields" value="14" alpha ...
[15] <paletteEntry color="#e68000" label="221 - Vineyards" value="15" alpha=" ...
[16] <paletteEntry color="#f2a64d" label="222 - Fruit trees and berry plantat ...
[17] <paletteEntry color="#e6a600" label="223 - Olive groves" value="17" alph ...
[18] <paletteEntry color="#e6e64d" label="231 - Pastures" value="18" alpha="2 ...
[19] <paletteEntry color="#ffe6a6" label="241 - Annual crops associated with ...
[20] <paletteEntry color="#ffe64d" label="242 - Complex cultivation patterns" ...
...colors = xml_attr(nodes, "color")
head(colors)[1] "#e6004d" "#ff0000" "#cc4df2" "#cc0000" "#e6cccc" "#e6cce6"labels = xml_attr(nodes, "label")
head(labels)[1] "111 - Continuous urban fabric"
[2] "112 - Discontinuous urban fabric"
[3] "121 - Industrial or commercial units"
[4] "122 - Road and rail networks and associated land"
[5] "123 - Port areas"
[6] "124 - Airports" legend_df = data.frame(Kolor = colors, Etykieta = labels)
head(legend_df) Kolor Etykieta
1 #e6004d 111 - Continuous urban fabric
2 #ff0000 112 - Discontinuous urban fabric
3 #cc4df2 121 - Industrial or commercial units
4 #cc0000 122 - Road and rail networks and associated land
5 #e6cccc 123 - Port areas
6 #e6cce6 124 - AirportsFormat JSON (ang. JavaScript Object Notation) jest formatem tekstowym bazującym na języku JavaScript, ale jest niezależny od tego języka.
library(jsonlite)
miasta = fromJSON("data/miasta.geojson")
miasta_df = miasta$features
str(miasta_df)'data.frame': 2 obs. of 3 variables:
$ type : chr "Feature" "Feature"
$ geometry :'data.frame': 2 obs. of 2 variables:
..$ type : chr "Point" "Point"
..$ coordinates:List of 2
.. ..$ : num 16.9 52.4
.. ..$ : num 22.6 51.3
$ properties:'data.frame': 2 obs. of 1 variable:
..$ name: chr "Poznań" "Lublin"miasta_df$longitude = sapply(miasta$features$geometry$coordinates,
function(x) x[[1]])
miasta_df$latitude = sapply(miasta$features$geometry$coordinates,
function(x) x[[2]])
miasta_df$geometry = NULL
miasta_df type name longitude latitude
1 Feature Poznań 16.92517 52.40836
2 Feature Lublin 22.57222 51.25564API (ang. Application Programming Interface) to zestaw reguł i protokołów, które umożliwiają komunikację między różnymi aplikacjami. W kontekście analizy danych, API są często wykorzystywane do pobierania danych z różnych źródeł, takich jak serwisy internetowe, bazy danych, czy aplikacje mobilne czy też komunikacji między różnymi aplikacjami. Mogą być one publiczne, prywatne, czy też zabezpieczone za pomocą kluczy API.1
library(httr2)
library(jsonlite)Adres konkretnego API składa się z dwóch części: adresu bazowego (ang. base URL) oraz końcówki (ang. endpoint). API najczęściej zwracają dane w formacie JSON, ale mogą także zwracać dane w formacie XML, CSV, czy innych.
https://www.weather.gov/documentation/services-web-api
base_url = "https://api.weather.gov/"
wsp = c(39.7456, -97.0892)
wsp = paste(wsp, collapse = ",")
endpoint_wsp = paste0("points/", wsp)
req_wsp = request(base_url) |>
req_url_path_append(endpoint_wsp)
req_perform_wsp = req_wsp |>
req_perform()
results_wsp = req_perform_wsp |>
resp_body_json()
results_wsp$properties$forecast[1] "https://api.weather.gov/gridpoints/TOP/32,81/forecast"results_weather = results_wsp$properties$forecast |>
request() |>
req_perform() |>
resp_body_json()
results_weather$properties$periods[[1]]$temperature[1] 45W wielu sytuacjach oprócz wczytywania danych z plików, baz danych, czy API, konieczne jest ręczne generowanie i wprowadzanie danych. W przypadku małych zbiorów danych można to zrobić po prostu poprzez wpisanie ich w kodzie. Natomiast w przypadku większych zbiorów danych, przydać się mogą funkcje, które generują określone wartości, takie jak seq(), rep(), czy sample().
Funkcja seq() generuje sekwencję liczb od pierwszej do ostatniej. Ma ona trzy warianty.
seq(1, 365, by = 7) [1] 1 8 15 22 29 36 43 50 57 64 71 78 85 92 99 106 113 120 127
[20] 134 141 148 155 162 169 176 183 190 197 204 211 218 225 232 239 246 253 260
[39] 267 274 281 288 295 302 309 316 323 330 337 344 351 358 365seq(1, 365, length.out = 53) [1] 1 8 15 22 29 36 43 50 57 64 71 78 85 92 99 106 113 120 127
[20] 134 141 148 155 162 169 176 183 190 197 204 211 218 225 232 239 246 253 260
[39] 267 274 281 288 295 302 309 316 323 330 337 344 351 358 365seq(1, 365, along.with = 1:53) [1] 1 8 15 22 29 36 43 50 57 64 71 78 85 92 99 106 113 120 127
[20] 134 141 148 155 162 169 176 183 190 197 204 211 218 225 232 239 246 253 260
[39] 267 274 281 288 295 302 309 316 323 330 337 344 351 358 365Funkcja rep() powtarza wartości i również ma trzy warianty.
rep(c("Poznań", "Kraków", "Warszawa"), times = 3)[1] "Poznań" "Kraków" "Warszawa" "Poznań" "Kraków" "Warszawa" "Poznań"
[8] "Kraków" "Warszawa"rep(c("Poznań", "Kraków", "Warszawa"), each = 3)[1] "Poznań" "Poznań" "Poznań" "Kraków" "Kraków" "Kraków" "Warszawa"
[8] "Warszawa" "Warszawa"rep(c("Poznań", "Kraków", "Warszawa"), length.out = 9)[1] "Poznań" "Kraków" "Warszawa" "Poznań" "Kraków" "Warszawa" "Poznań"
[8] "Kraków" "Warszawa"Ostatnia podstawowa technika generowania danych to stosowanie metod losowych. R posiada szereg funkcji generujących liczby losowe z różnych rozkładów teoretycznych, takich jak runif(), rnorm(), czy rbinom(). Inna przydatna funkcja to sample(), która losowo wybiera wartości z zadanego wektora. Domyślnie funkcja sample() losuje bez zwracania, ale można to zmienić za pomocą argumentu replace = TRUE.
sample(1:5, size = 5)[1] 2 3 4 5 1sample(1:5, size = 3)[1] 4 5 3sample(1:5, size = 10)Error in sample.int(length(x), size, replace, prob): cannot take a sample larger than the population when 'replace = FALSE'sample(1:5, size = 10, replace = TRUE) [1] 4 4 2 5 5 2 1 5 3 2Należy pamiętać, że funkcje generujące liczby losowe będą zwracać różne wartości za każdym razem, gdy zostaną wywołane. Aby uzyskać te same wartości, należy ustawić ziarno generatora liczb losowych za pomocą funkcji set.seed() przed wywołaniem funkcji generujących liczby losowe.
Isnieje wiele innych formatów danych, różniących się pod względem struktury czy zastosowania. Ciągle także tworzone są nowe formaty. Wielowymiarowe dane są często przechowywane w formatach, takich jak NetCDF, HDF5, czy GRIB, a współcześnie także w formatach takich jak Zarr. Innym przykładem jest zdobywająca popularność grupa formatów związanych z danymi ułożonymi kolumnami, takich jak Arrow czy Parquet. Ich głównym założeniem jest optymalizacja operacji takich jak analiza danych, uczenie maszynowe, czy przetwarzanie strumieniowe.
Pliki binarne są plikamii, które przechowują dane w postaci ciągów bitów bezpośrednio zrozumiałym przez komputer. Są one zazwyczaj szybsze i zajmują mniej miejsca niż pliki tekstowe, ale są mniej czytelne dla człowieka. Można je w R odczytywać i zapisywać za pomocą funkcji readBin() i writeBin().
con = file("data/binary_file.bin", "rb")
data = readBin(con, "numeric", n = 1, size = 8) # size zależy on typu zapisanych danych
data[1] 1.23close(con)Przed wykonaniem analizy i wizualizacji danych, zazwyczaj konieczne jest oczyszczenie i poprawienie danych. Część tych aspektów, jak na przykład przekształcenie formy danych, była już omówiona wcześniej. Tutaj rozważmy inne kwestie.
Pierwsza z nich to nazwy kolumn. Z perspektywy człowieka, nazwy kolumn powinny być proste, zrozumiałe i czytelne. Dodatkowo, w przypadku zmiennych posiadających konkretną jednostkę, warto dodać ją do nazwy kolumny. Z drugiej strony, z perspektywy komputera, nazwy kolumn nie powinny zawierać spacji, polskich znaków, ani znaków specjalnych. Czesto też należy unikać rozpoczyania nazw kolumn od cyfr. Przykładowo, zobaczmy kolejne możliwe nazwy kolumny: 1C -> C -> c -> ciśnienie -> cisnienie -> cisnienie_hPa.
Kolejnym aspektem jest poprawianie typów danych dla kolumn. W przypadku danych liczbowych, warto sprawdzić, czy nie zawierają one białych znaków, znaków specjalnych, czy innych niepożądanych znaków. Po usunięciu tych znaków należy pamiętać o zmianie typu danych na numeryczny.
a = c(1, 2, 3, " 4", 5)
a[1] "1" "2" "3" " 4" "5" mean(a)[1] NAb = stringr::str_trim(a)
b[1] "1" "2" "3" "4" "5"mean(b)[1] NAd = as.numeric(b)
d[1] 1 2 3 4 5mean(d)[1] 3Następny aspekt obejmuje poprawianie pojedynczych wartości zmiennych.2
a = c("dobrze", "poprawnie", "źle", "abrdzo źle")
a[1] "dobrze" "poprawnie" "źle" "abrdzo źle"b = ifelse(a == "abrdzo źle", "bardzo źle", a)
b[1] "dobrze" "poprawnie" "źle" "bardzo źle"Ostatni główny aspekt to dodawanie nowych zmiennych na podstawie istniejących. Tutaj przykładowo możemy stworzyć nową zmienną logiczną na podstawie wartości innej zmiennej, stworzyć zmienną o typie daty na podstawie innych zmiennych reprezentujących dzień, miesiąc i rok, czy też stworzyć zmienną z wartościami kategorycznymi na podstawie wartości numerycznych.
# 1
a1 = c(1, 2, 3, 4, 5)
a1[1] 1 2 3 4 5a2 = ifelse(a1 > 3, TRUE, FALSE)
a2[1] FALSE FALSE FALSE TRUE TRUE# 2
dzien = c(1, 2, 3, 4, 5)
miesiac = c(1, 1, 2, 2, 3)
rok = c(2023, 2023, 2023, 2023, 2023)
data = as.Date(paste(rok, miesiac, dzien, sep = "-"))
data[1] "2023-01-01" "2023-01-02" "2023-02-03" "2023-02-04" "2023-03-05"# 3
b1 = c(1, 2, 3, 4, 5)
b1[1] 1 2 3 4 5b2 = dplyr::case_when(
b1 < 2 ~ "niski",
b1 < 4 ~ "średni",
b1 >=4 ~ "wysoki"
)
b2[1] "niski" "średni" "średni" "wysoki" "wysoki"Inne bardziej zaawansowane metody poprawiania danych dotyczą zrozumienia oraz imputacji brakujących wartości.
Przykłady łączenia danych zostaną pokazane na przykładzie pięciu zbiorów danych.
library(dplyr)Pierwszy z nich zawiera dane dotyczące populacji, oczekiwanej długości życia, oraz PKB na mieszkańca dla różnych krajów w różnych regionach w latach 1950-2023.
glimpse(gm1)Rows: 14,060
Columns: 6
$ Kraj <chr> "Australia", "Australia", "Australia", "Australia", "Aust…
$ Rok <dbl> 1950, 1951, 1952, 1953, 1954, 1955, 1956, 1957, 1958, 195…
$ Region <chr> "Azja", "Azja", "Azja", "Azja", "Azja", "Azja", "Azja", "…
$ Populacja <dbl> 8180000, 8420000, 8630000, 8820000, 9000000, 9210000, 943…
$ PKB_na_osobe <dbl> 16400, 16500, 16200, 16500, 17100, 17600, 17600, 17500, 1…
$ Ocz_dl_zycia <dbl> 69.4, 69.2, 69.5, 69.9, 70.2, 70.3, 70.4, 70.6, 71.0, 70.…Drugi zbiór danych zawiera informacje o krajach, latach, oraz wartościach CO_2_ na osobę i płodności dla tych samych krajów.
glimpse(gm2)Rows: 14,060
Columns: 4
$ Kraj <chr> "Australia", "Australia", "Australia", "Australia", "Aust…
$ Rok <dbl> 1950, 1951, 1952, 1953, 1954, 1955, 1956, 1957, 1958, 195…
$ CO2_na_osobe <dbl> 6.70, 7.04, 7.01, 6.77, 7.57, 7.72, 7.80, 7.78, 7.92, 8.3…
$ Plodnosc <dbl> 3.07, 3.06, 3.18, 3.19, 3.20, 3.28, 3.33, 3.42, 3.42, 3.4…Trzeci zbiór to podzbiór pierwszego zbioru danych dla roku 2019.
gm1_2019 = filter(gm1, Rok == 2019)
glimpse(gm1_2019)Rows: 190
Columns: 6
$ Kraj <chr> "Australia", "Brunei", "Kambodża", "Chiny", "Fidżi", "Hon…
$ Rok <dbl> 2019, 2019, 2019, 2019, 2019, 2019, 2019, 2019, 2019, 201…
$ Region <chr> "Azja", "Azja", "Azja", "Azja", "Azja", "Azja", "Azja", "…
$ Populacja <dbl> 2.54e+07, 4.38e+05, 1.62e+07, 1.42e+09, 9.18e+05, 7.50e+0…
$ PKB_na_osobe <dbl> 49400, 61400, 4460, 16000, 13200, 59600, 11900, 41700, 19…
$ Ocz_dl_zycia <dbl> 82.9, 74.4, 69.9, 77.6, 68.4, 84.5, 71.4, 84.8, 60.8, 73.…Czwarty zbiór to podzbiór drugiego zbioru danych dla roku 2019.
gm2_2019 = filter(gm2, Rok == 2019)
glimpse(gm2_2019)Rows: 190
Columns: 4
$ Kraj <chr> "Australia", "Brunei", "Kambodża", "Chiny", "Fidżi", "Hon…
$ Rok <dbl> 2019, 2019, 2019, 2019, 2019, 2019, 2019, 2019, 2019, 201…
$ CO2_na_osobe <dbl> NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, NA, N…
$ Plodnosc <dbl> 1.82, 1.84, 2.47, 1.64, 2.45, 1.36, 2.29, 1.50, 3.53, 1.8…Piąty zbiór to podzbiór pierwszego zbioru danych dla regionu Europy w roku 2019.
gm3_2019 = filter(gm1, Rok == 2019, Region == "Europa")
glimpse(gm3_2019)Rows: 45
Columns: 6
$ Kraj <chr> "Albania", "Andora", "Armenia", "Austria", "Azerbejdżan",…
$ Rok <dbl> 2019, 2019, 2019, 2019, 2019, 2019, 2019, 2019, 2019, 201…
$ Region <chr> "Europa", "Europa", "Europa", "Europa", "Europa", "Europa…
$ Populacja <dbl> 2870000, 76300, 2820000, 8880000, 10200000, 9670000, 1150…
$ PKB_na_osobe <dbl> 13700, 56300, 14300, 55800, 14400, 19300, 52000, 14900, 2…
$ Ocz_dl_zycia <dbl> 78.5, 82.2, 75.7, 82.2, 71.0, 74.0, 81.4, 77.0, 73.3, 78.…Dane z wielu tabel można łączyć używając funkcji rbind() lub cbind(). Funkcja rbind() łączy dane według wierszy (dodaje wiersze), podczas gdy funkcja cbind() łączy dane według kolumn (dodaje kolumny). Jednakże, obie funkcje wymagają, by dane miały taką samą liczbę kolumn (dla rbind()) lub wierszy (dla cbind()).3
W bardziej złożonych przypadkach, gdy dane mają różną liczbę wierszy i kolumn, stosuje się złączenia. W porównaniu do funkcji rbind() i cbind(), złączenia pozwalają na łączenie danych z tabel o różnej liczbie wierszy i kolumn w oparciu o wspólną zmienną(e) (znaną również jako klucz). Istnieje kilka rodzajów złączeń, które możemy podzielić na dwie ogólne grupy: złączenia modyfikujące i złączenia filtrujące.
Złączenia modyfikujące to takie, które dodają nowe zmienne do jednej ramki danych na podstawie pasujących obserwacji w innej ramce.
| Kod_złączenia | Nazwa_złączenia | Opis |
|---|---|---|
| left_join() | Lewe | Wszystkie obserwacje z lewej ramki danych zostają zachowane, pasujące obserwacje z prawej ramki danych zostają dodane. |
| right_join() | Prawe | Wszystkie obserwacje z prawej ramki danych zostają zachowane, a pasujące obserwacje z lewej ramki danych zostają dodane. |
| inner_join() | Wewnętrzne | Zachowane są tylko obserwacje, które pasują do obu ramek danych. |
| full_join() | Pełne | Zachowane są wszystkie obserwacje, a brakujące wartości są uzupełniane jako NA. |

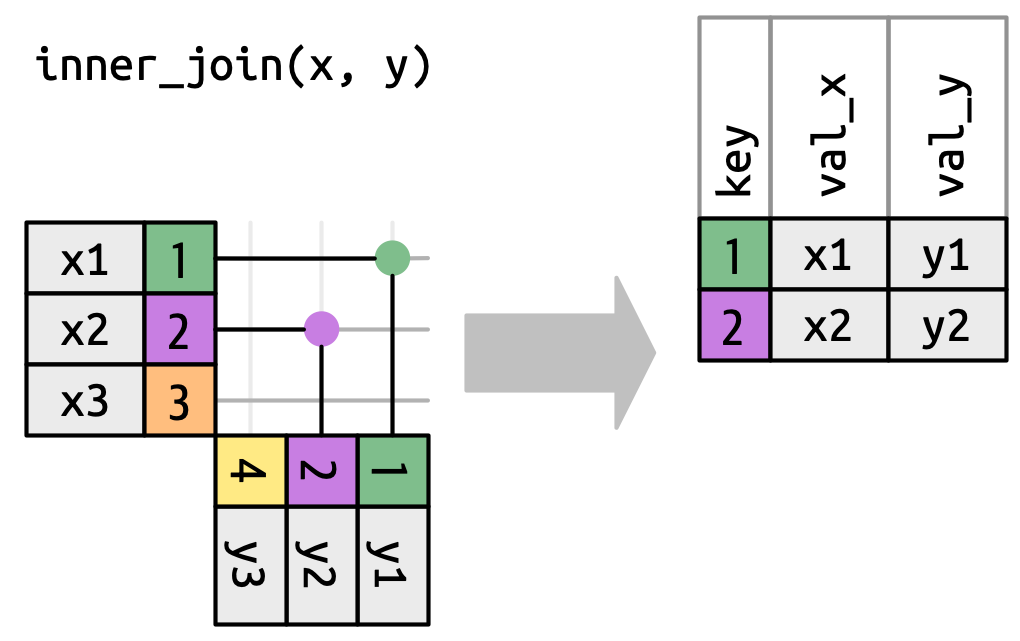
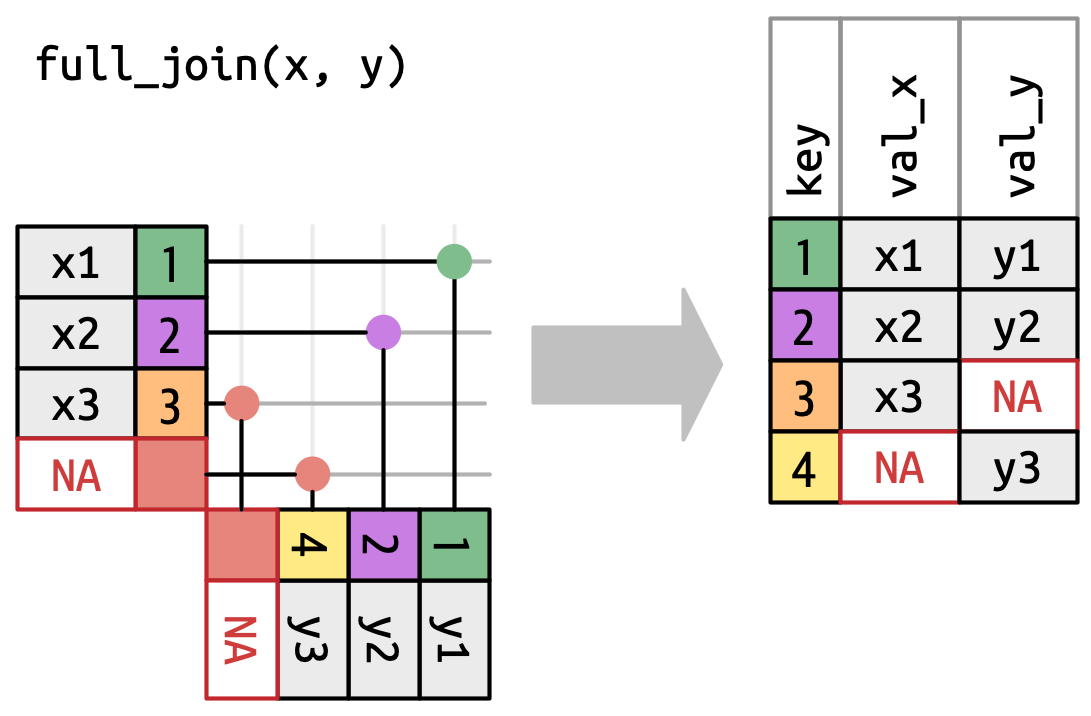
Wickham, Mine Cetinkaya-Rundel, i Grolemund (2023)
Złączenia lewe (ang. left joins) zachowują wszystkie obserwacje z lewej ramki danych, dodając odpowiadające obserwacje z prawej ramki danych na podstawie podanego klucza. Obserwacje, które nie mają pasujących obserwacji w prawej ramce danych, otrzymują wartości NA.
Kluczem może być jedna lub więcej zmiennych (kolumn). W przypadku, gdy te kolumny mają taką samą nazwę wystarczy wskazać ją jako argument funkcji by.
gm_2019_lj = left_join(gm1_2019, gm2_2019, by = "Kraj")
gm_2019_lj# A tibble: 190 × 9
Kraj Rok.x Region Populacja PKB_na_osobe Ocz_dl_zycia Rok.y CO2_na_osobe
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Australia 2019 Azja 25400000 49400 82.9 2019 NA
2 Brunei 2019 Azja 438000 61400 74.4 2019 NA
3 Kambodża 2019 Azja 16200000 4460 69.9 2019 NA
# ℹ 187 more rows
# ℹ 1 more variable: Plodnosc <dbl>Możliwe jest również złączenie na podstawie wielu kolumn.
gm_2019_lj2 = left_join(gm1_2019, gm2_2019, by = c("Kraj", "Rok"))
gm_2019_lj2# A tibble: 190 × 8
Kraj Rok Region Populacja PKB_na_osobe Ocz_dl_zycia CO2_na_osobe Plodnosc
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Austra… 2019 Azja 25400000 49400 82.9 NA 1.82
2 Brunei 2019 Azja 438000 61400 74.4 NA 1.84
3 Kambod… 2019 Azja 16200000 4460 69.9 NA 2.47
# ℹ 187 more rowsGdy klucze mają różne nazwy, można użyć funkcji join_by() w argumencie by. Przykładowo, by = join_by("Kraj" = "Country") oznacza, że kolumna Kraj z lewej ramki danych jest łączona z kolumną Country z prawej ramki danych.
Złączenia prawe (ang. right joins) zachowują wszystkie obserwacje z prawej ramki danych, dodając odpowiadające obserwacje z lewej ramki danych na podstawie podanego klucza.
gm_2019_rj = right_join(gm1_2019, gm2_2019, by = "Kraj")
gm_2019_rj# A tibble: 190 × 9
Kraj Rok.x Region Populacja PKB_na_osobe Ocz_dl_zycia Rok.y CO2_na_osobe
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Australia 2019 Azja 25400000 49400 82.9 2019 NA
2 Brunei 2019 Azja 438000 61400 74.4 2019 NA
3 Kambodża 2019 Azja 16200000 4460 69.9 2019 NA
# ℹ 187 more rows
# ℹ 1 more variable: Plodnosc <dbl>Złączenia wewnętrzne (ang. inner joins) zachowują tylko obserwacje, które znajdują się w zmiennej klucza(y) obu ramek danych.
gm_2019_ij = inner_join(gm1_2019, gm2_2019, by = "Kraj")
gm_2019_ij# A tibble: 190 × 9
Kraj Rok.x Region Populacja PKB_na_osobe Ocz_dl_zycia Rok.y CO2_na_osobe
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Australia 2019 Azja 25400000 49400 82.9 2019 NA
2 Brunei 2019 Azja 438000 61400 74.4 2019 NA
3 Kambodża 2019 Azja 16200000 4460 69.9 2019 NA
# ℹ 187 more rows
# ℹ 1 more variable: Plodnosc <dbl>Złączenia pełne (ang. full joins) zachowują wszystkie obserwacje z obu ramek danych, a brakujące wartości są uzupełniane jako NA.
gm_2019_fj = full_join(gm1_2019, gm2_2019, by = "Kraj")
gm_2019_fj# A tibble: 190 × 9
Kraj Rok.x Region Populacja PKB_na_osobe Ocz_dl_zycia Rok.y CO2_na_osobe
<chr> <dbl> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Australia 2019 Azja 25400000 49400 82.9 2019 NA
2 Brunei 2019 Azja 438000 61400 74.4 2019 NA
3 Kambodża 2019 Azja 16200000 4460 69.9 2019 NA
# ℹ 187 more rows
# ℹ 1 more variable: Plodnosc <dbl>Druga grupa złączeń to złączenia filtrujące, które wybierają obserwacje z jednej ramki danych na podstawie tego, czy odpowiadają one do obserwacji w innej ramce.
| Kod_złączenia | Nazwa_złączenia | Opis |
|---|---|---|
| semi_join | Pół-złączenie | Zachowane są tylko obserwacje, które pasują do obu ramek danych. |
| anti_join | Anty-złączenie | Zachowane są tylko obserwacje, które nie pasują do obu ramek danych. |
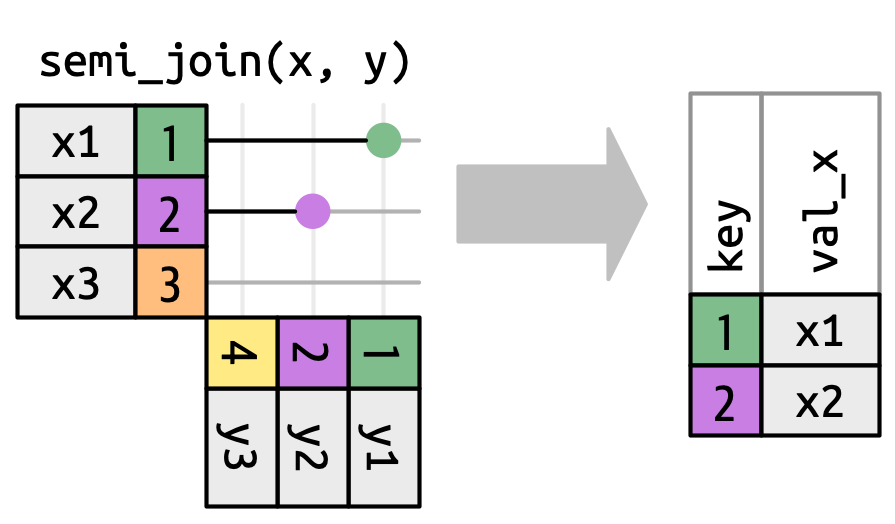
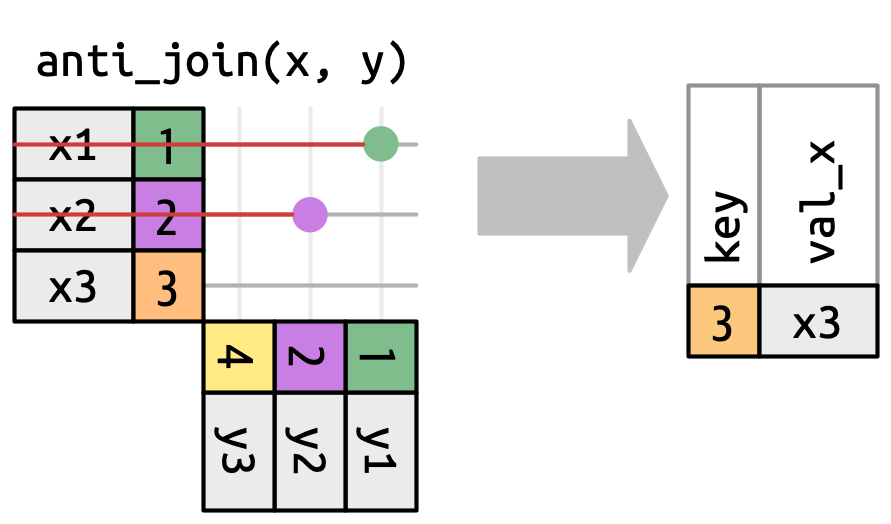
Wickham, Mine Cetinkaya-Rundel, i Grolemund (2023)
Pół-złączenia (ang. semi joins) zachowują tylko obserwacje, które pasują do obu ramek danych. W przeciwieństwie jednak od np. wewnętrznych złączeń, pół-złączenia zachowują tylko kolumny z lewej ramki danych.
gm_2019_sj = semi_join(gm1_2019, gm3_2019, by = "Kraj")
gm_2019_sj# A tibble: 45 × 6
Kraj Rok Region Populacja PKB_na_osobe Ocz_dl_zycia
<chr> <dbl> <chr> <dbl> <dbl> <dbl>
1 Albania 2019 Europa 2870000 13700 78.5
2 Andora 2019 Europa 76300 56300 82.2
3 Armenia 2019 Europa 2820000 14300 75.7
# ℹ 42 more rowsAnty-złączenia (ang. anti joins) zachowują tylko obserwacje, które nie pasują do obu ramek danych. W efekcie anty-złączenia otrzymuje się tylko kolumny z lewej ramki danych.
gm_2019_aj = anti_join(gm1_2019, gm3_2019, by = "Kraj")
gm_2019_aj# A tibble: 145 × 6
Kraj Rok Region Populacja PKB_na_osobe Ocz_dl_zycia
<chr> <dbl> <chr> <dbl> <dbl> <dbl>
1 Australia 2019 Azja 25400000 49400 82.9
2 Brunei 2019 Azja 438000 61400 74.4
3 Kambodża 2019 Azja 16200000 4460 69.9
# ℹ 142 more rowslibrary(readr)
write_csv(gm, "data/gm2.csv")library(writexl)
write_xlsx(list(gapminder = gm), "data/gm2.xlsx")library(DBI)
library(readr)
library(RSQLite)
conn = dbConnect(RSQLite::SQLite(), "data/gapminder_1950_2023.sqlite")
dbWriteTable(conn, "gapminder_1950_2023", gm)
dbDisconnect(conn)library(xml2)
library(tibble)
lc_legend = data.frame(
Kolor = c("#e6004d", "#ff0000", "#cc4df2", "#cc0000", "#e6cccc", "#e6cce6"),
Etykieta = c("111 - Continuous urban fabric", "112 - Discontinuous urban fabric",
"121 - Industrial or commercial units",
"122 - Road and rail networks and associated land",
"123 - Port areas", "124 - Airports")
)
root = xml_new_document() |>
xml_add_child("root")
for(i in 1:nrow(lc_legend)) {
row = xml_add_child(root, "row")
xml_add_child(row, "Kolor", lc_legend$Kolor[i])
xml_add_child(row, "Etykieta", lc_legend$Etykieta[i])
}
write_xml(root, "lc_legend.xml")Takiego klucza nie powinno się przekazywać dalej (np. wewnątrz skryptu), a zamiast tego przechowywać go w bezpiecznym miejscu, takim jak zmienna środowiskowa. Takie zmienne można ustawić, np. za pomocą pakietu keyring.↩︎
Tutaj pomocna może być także funkcja dplyr::if_else().↩︎
Alternatywnie, można użyć funkcji bind_rows() z pakietu dplyr, która działa także gdy w obu zbiorach danych część kolumn jest różna.↩︎